김빛내리 IBS 단장, 드로셔 단백질 기능 규명
IBS(기초과학연구원·원장 김두철)는 김빛내리 RNA연구단장 연구팀이 마이크로RNA가 드로셔 단백질에 의해 형성되는 과정을 인체 세포 내에서 확인하는데 성공했다고 23일 밝혔다.
연구팀은 드로셔 단백질이 마이크로RNA를 자르는 위치를 정확하게 분석할 수 있는 새로운 실험법을 개발했다. 이 분석법을 통해 그동안 알려져 있던 마이크로RNA 정보가 일부 부정확했음을 밝히고 드로셔와 마이크로RNA 각각의 새로운 특성들을 규명했다.
드로셔 단백질은 마이크로RNA 1차 전구체를 특정 위치에서 자르는 효소다. 지난 2003년 김빛내리 단장 연구팀에 의해 마이크로RNA 생성에 필수적인 효소로 밝혀진 바 있다.
연구팀은 드로셔 단백질의 3차원 구조 규명에 주안점을 두고 드로셔가 마이크로RNA 1차 전구체의 어느 부위를 인식해 절단하는지에 관한 패턴을 일반화하는데 성공했다.
드로셔 구조와 일반적인 RNA 절단 패턴은 밝혀졌지만, 실제 인체 세포 내에서 드로셔에 의해 만들어지는 마이크로RNA 각각에 관한 정보는 부족했다. 드로셔가 마이크로RNA 생성을 주도하는 역할 외에 어떠한 기능이 있는지도 불명확했다.
연구팀은 자외선이 아닌 포름알데히드를 이용하는 방식인 f-클립시크로 실험법을 대체했다. 포름알데히드는 RNA와 드로셔의 결합 부위에 끼어 들어가 둘을 이어준다. 이 상태에서 드로셔에 특이적으로 결합하는 항체를 처리하면 항원-항체반응으로 드로셔만을 모아 드로셔에 붙어있는 RNA 산물을 염기서열 분석법으로 읽어낼 수 있다.
연구팀은 사람의 배아 신장 세포와 자궁경부암 세포 두 종류를 대상으로 f-클립시크를 적용해 수백개에 달하는 마이크로RNA 전구체 상 드로셔 절단 위치를 찾는데 성공했다.
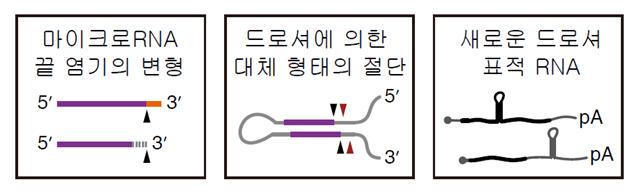
이번 실험에서 얻은 마이크로RNA 데이터를 세계 최대의 마이크로RNA 데이터베이스인 '미르베이스(miRBase)'와 비교한 결과, 드로셔가 만드는 마이크로RNA 중 상당수가 불일치함을 확인했다.
미르베이스는 생성이 완료된 마이크로RNA만을 기준으로 하는데, 많은 마이크로RNA가 생성되는 도중에 염기서열의 끝부분에서 추가적인 변형이 일어나기 때문으로 분석됐다.
또 동일한 마이크로RNA 전구체라도 드로셔에 의해 각각 다른 부위가 절단돼 여러 종류의 마이크로RNA가 만들어질 수 있는데 극소수의 마이크로RNA에서만 이러한 현상이 보고됐던 것과 달리 이번 연구를 통해 실제로는 다수의 마이크로RNA에서 일어난다는 사실을 증명했다.
드로셔는 RNA를 잘라 신경 발생이나 골수 형성 과정을 조절할 뿐만 아니라 바이러스 증식을 억제하는 기능도 있는 것으로 보고돼 왔다.
김빛내리 단장은 "이번 연구는 드로셔가 결합하는 RNA를 정확히 찾아낼 수 있는 연구 방법을 제공함으로써, 생물학적으로 중요한 여러 현상들에서 드로셔의 기능을 규명할 길을 열 것"이라며 "이번 연구대상 외의 다른 인체 세포에서도 드로셔에 의해 만들어지는 마이크로RNA 정보를 밝혀나갈 계획"이라고 말했다.
이번 연구결과는 셀(Cell)의 자매지인 '몰레큘러 셀(Molecular Cell)'에 미국 동부시간으로 20일자로 게재됐다.
댓글 정렬