생명연, 병원균 DB 'PAIDB 2.0' 개발
미생물 병원성·항생제 내성 유무 분석 가능
지금까지 알려진 모든 병원성 유전자 부위와 항생제 내성 유전자 부위의 빠른 검색과 분석이 가능한 시스템이 국내 연구진에 의해 개발됐다.
한국생명공학연구원(원장 오태광)은 윤성호 바이오합성연구센터 박사 연구팀이 다양한 병원균 내에 존재하는 감염병 관련 유전인자와 항생제 내성 유전인자를 모두 조사해 데이터베이스화 한 'PAIDB 2.0'를 개발해 공개했다고 22일 밝혔다.
'PAIDB 2.0'을 활용하면 병원성 및 항생제 내성 유전자를 검색하고 예측할 수 있다는 것이 연구팀의 설명이다.
미생물 유전체 안의 특정 유전자들은 다른 균주로 쉽게 이동할 수 있고 대부분 병원성 및 항생제 내성 유전자를 포함하고 있어 신·변종 병원균의 출현하거나 감염병·동식물 질병 확산으로 이어진다. 또 항생제 내성을 가지게 되면 슈퍼박테리아(다제내성균)이 출현하게 된다.
다른 미생물로부터 도입된 병원균의 병원성 유전자 부위와 항생제 내성 유전자 부위는 각각 병원성 유전자 '섬'(pathogenicity island), 내성 유전자 '섬' (resistance island)이라고 불리는데, 연구팀은 지금까지 알려진 1331개의 병원성 유전자섬과 108개의 내성 유전자 섬을 조사했다.
아울러 2673종의 미생물 유전체를 분석해 1596개의 병원성 유전자군과 210개의 항생제 내성 유전자군을 예측했다.
이를 바탕으로 병원성 유전자군 및 내성 유전자군을 탐지할 수 있는 검색엔진을 업그레이드하고 기존 미생물에 새로운 병원성 및 항생제 내성 유무를 분석할 수 있게 했다.
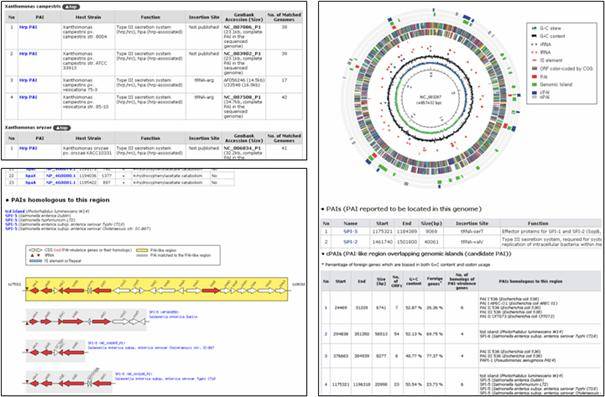
연구를 주도한 윤성호 박사는 지난 2005년 다양한 미생물 균주의 유전체 내에서 병원성 유전자 군을 컴퓨터상에서 찾아낼 수 수 있는 알고리즘을 개발했고, 다음해 병원균의 병원성 유전자 데이터베이스인 PAIDB를 처음 공개했다. 이번에 개발된 PAIDB 2.0은 지금까지 알려진 항생제 내성 유전자군의 정보도 추가하였으며, 병원성 및 저항성 유전자군의 검색 시스템을 강화 했다는 것이 연구팀의 설명이다.
오태광 원장은 "현재 전 세계적으로 신·변종 감염병, 바이오테러, 생화학전, 동식물 질병의 확산과 슈퍼박테리아의 출현이 큰 문제로 대두되는 상황에서, 본 시스템의 구축은 국가재난성 감염병 및 슈퍼박테이아에 대한 진단, 예방기술 개발에 기여할 수 있을 것"이라고 평가했다.
이번 연구는 미래창조과학부와 한국연구재단이 시행하는 기후변화대응 기술개발사업과 생명연 전문연구사업의 지원을 받아 수행됐고, 연구개발성과는 생명과학 분야 전문저널인 Nucleic Acids Research의 Advance Access에 21일자로 공개됐고 2015년 1월 데이터베이스 이슈에 정식논문으로 게재될 예정이다.
PAIDB 2.0은 PAIDB 웹사이트 (http://www.paidb.re.kr)에서 무료로 이용할 수 있다.
댓글 정렬