첨단 텍스트마이닝 기술 검색엔진 '온코서치' 개발
유전자 역할 표헌 없어도 연구문헌·DB 검색 가능
암은 수천개 이상 유전자의 비정상적인 변화와 그에 따른 신호전달 체계 교란이 주요 원인이다. 따라서 암을 이해하고 치료하기 위해서는 이들 유전자의 변화에 대한 정보를 파악하고, 수집·분석하는 일이 중요하다.
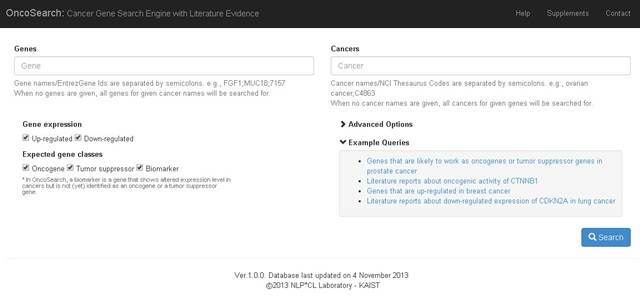
국내 연구진이 암 관련 유전자 정보를 보다 빠르고 정확하게 찾을 수 있는 검색 엔진을 개발했다.
박종철 KAIST(한국과학기술원·총장 강성모) 전산학과 연구팀과 이현주 GIST(광주과학기술원·총장 김영준) 교수는 언어학, 컴퓨터공학, 생물학, 의학을 포괄적으로 연계한 융합연구를 통해 암 관련 유전자 정보 검색 엔진 '온코서치(OncoSearch)'를 개발했다.
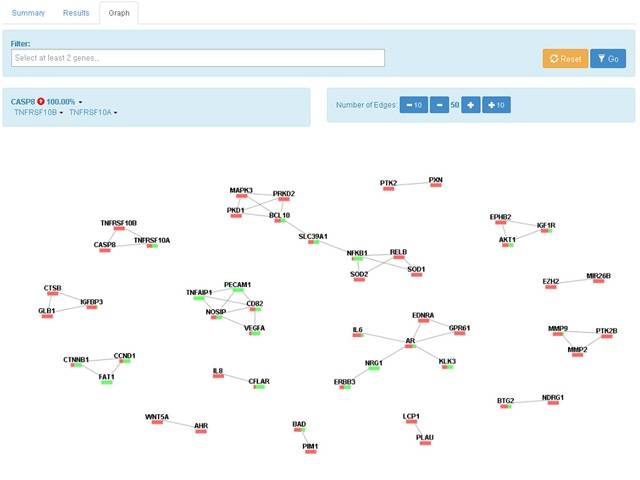
이번 연구로 생물학과 의학 연구문헌 데이터베이스인 '메드라인(Medline)'에 탑재된 300만 건의 자료 가운데서 유전자의 발현량 변화와 유전자 변화에 따른 암 상태 변화를 기술하는 문장을 보다 쉽게 찾아낼 수 있게 됐다.
온코서치는 첨단 텍스트마이닝 기술인 사건 정보 추출시스템과 최대 엔트로피 분류기를 사용해 문장의 구조를 분석한다. 특히 암 관련 유전자 역할에 대한 표현(oncogene, tumor suppressor)이 없어도 관련된 정보를 파악할 수 있다.
박종철 교수는 "온코서치가 연구문헌에서 자동으로 수집한 암 과련 유전자에 대한 정보는 앞으로 자동 추론기술 등을 활용해 암 연구를 위한 새로운 도구로 활용 될 것"이라고 기대했다.
온코서치는 현재 웹페이지(http://oncosearch.biopathway.org)에 접속해 사용이 가능하다.
댓글 정렬