조광현 KAIST 교수 연구팀, 5개 마이오마커 유전자 확인
KAIST(총장 신성철)는 조광현 바이오및뇌공학과 교수 연구팀이 대장암의 항암제 내성을 극복할 수 있는 새로운 병용 치료 타겟을 발굴하는데 성공했다고 7일 밝혔다.
대장암은 대표적인 난치암으로 알려진다. 국내에서도 서구화된 식습관과 비만으로 대장암 발병률 속도가 지난 10년간 가파르게 증가하는 추세다.
암치료제로 암세포의 특정 분자만을 표적하는 표적항암제가 개발되면서 부작용을 크게 줄이고 효과를 높일 수 있게 됐다. 하지만 약물에 반응하는 환자가 제한적이고 반응을 보이더라도 표적 항암치료 후 약물 내성으로 암이 재발하는 문제를 안고 있다.
또 환자별로 항암제에 대한 반응이 달라 환자의 암 조직 내 유전자 변이 특징에 적합하게 치료하는 정밀의학의 필요성이 커지고 있다.
대장암도 약물 효과를 예측할 수 있는 유전자 바이오마커 여부에 따라 적합한 표적 항암제를 처방하는 시도가 이뤄진다. FDA 승인을 받은 대표적인 대장암 치료제인 세툭시맙(cetuximab). 약물 반응성을 예측하는 바이오마커로 KRAS 유전자 돌연변이의 유무가 관건이다. 유전자 돌연변이가 없는 환자에게 처방을 권고한다.
하지만 KRAS 돌연변이가 없는 환자도 세툭시맙 반응률이 절반 정도. 기존 항암 화학요법 단독시행과 비교해도 평균 5개월의 수명을 연장하는데 그치고 있다.
때문에 KRAS 돌연변이 유무 이외의 새 바이오마커가 요구되고 돌연변이가 존재해도 내성을 극복할 수 있는 병용치료 타겟 발굴이 필요하다.
연구팀은 암세포의 복잡한 생체데이터를 분자 네트워크 관점에서 분석하는 시스템생물학 접근법의 중요성을 제시했다.
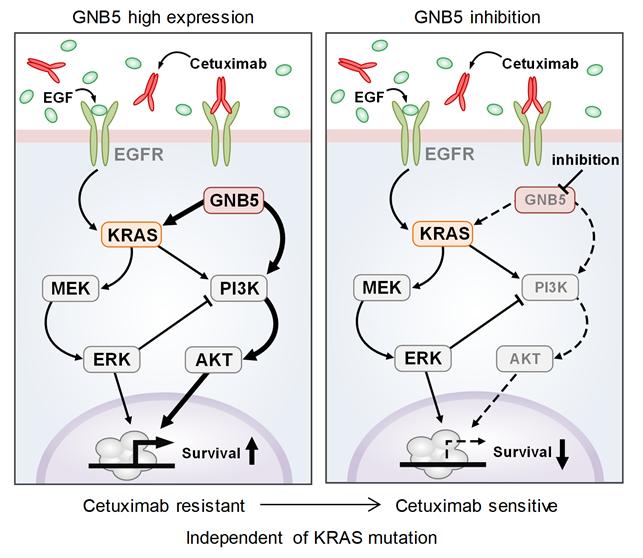
우선 연구팀은 유전체 데이터 분석, 수학 모델링, 컴퓨터 시뮬레이션 분석과 암 세포주 실험을 융합한 시스템생물학 연구를 통해 다섯개의 새로운 유전자(DUSP4, ETV5, GNB5, NT5E, PHLDA1)를 세툭시맙 반응 바이오마커로 찾아냈다.
각 유전자는 실험적으로 억제한 결과 KRAS 정상세포에서 발생하는 세툭시맙 내성을 모두 극복했다. 특히 GNB5를 억제하면 KRAS 돌연변이가 있는 세포주에서도 세툭시맙 처리에 따른 약물내성을 극복하는 것으로 확인됐다.
이번 성과로 암세포가 가진 약제 내성의 원리를 시스템 차원에서 파악하고 새로운 약물 타겟을 체계적으로 발굴할 것으로 기대된다. 또 발굴된 유전자들을 표적화하는 신약개발을 통해 내성을 가진 환자군에 대해서도 새로운 치료전략을 제시할 것으로 전망된다.
조 교수는 "지금껏 GNB5 유전자 조절을 대장암의 조합치료에 활용한 예는 없었다"며 "시스템 생물학으로 암세포가 가지는 약제 내성 원리를 밝히고 내성 환자군의 바이오마커 동정, 내성 극복을 위한 병행치료 타겟 발굴로 정밀의학 실현 가능성을 제시했다"고 연구 의의를 설명했다.
이번 연구는 과학기술정보통신부와 한국연구재단의 중견연구자지원사업, 바이오의료기술개발 사업의 지원을 받았다. 연구에는 박상민 박사과정, 황채영 박사 등이 참여했으며 결과는 국제학술지 '유럽생화학회저널(FEBS Journal)'의 4월호 표지논문으로 게재됐다.
댓글 정렬