이현주 교수 "향후 암 연관 유전변이 영역 찾는데 활용"
GIST(광주과학기술원·총장 문승현)는 이현주 전기전자컴퓨터공학부 교수 연구팀이 차세대 염기서열 데이터를 활용해 암과 연관성이 높은 유전변이 영역을 발굴하는 알고리즘을 개발했다고 10일 밝혔다.
차세대 염기서열 데이터란 유전체를 무수히 많은 짧은 길이의 DNA조각들로 나눈 뒤 병렬적인 서열분석을 통해 얻은 정보를 말한다.
연구팀은 먼저 개별 암 세포들로부터 획득한 차세대 염기서열 데이터 내 노이즈를 '웨이블릿 변환'이라는 수학적 기법을 활용해 제거하고 체세포 유전자의 유전자 개수가 변한 영역을 검출했다.
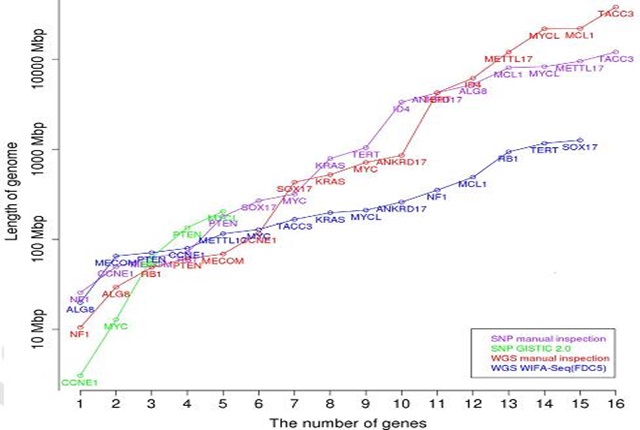
이현주 교수는 47개의 난소암 샘플에 개발한 알고리즘을 적용해 기존의 방법론보다 두 배 가까운 수의 암 연관 유전자를 찾아냄을 확인했다.
이 교수는 "이 알고리즘은 바이오 빅 데이터로부터 암과 연관된 유전변이 영역을 찾는 데 널리 활용될 수 있을 것으로 기대된다"고 말했다.
이번 연구결과는 국제학술지 '사이언티픽 리포츠(Scientific Reports)'의 지난 9일자에 게재됐다.
백승민 기자
bsm1702@hellodd.com
댓글 정렬